国家試験で出題される遺伝子解析法について解説します!最近出題頻度が増えてきている範囲なので,対策が必須になってくる?
各見出しの重要度は,過去の臨床検査技師国家試験(第52回~最新回まで)の出題率や覚えやすさなどを考慮して主観で表記しています。国家試験を受ける学生以外の方は無視してかまいません。
- 遺伝子解析法(重要度:★★★★★)
- PCR法(polymerase chain reaction)(重要度:★★★★★)
- リアルタイムPCR法(real-time polymerase chain reaction)(重要度:★★★☆☆)
- RT-PCR法(reverse transcription polymerase chain reaction)(重要度:★★★★☆)
- LAMP法(loop mediated isothermal amplification)(重要度:★★☆☆☆)
- TMA法(transcription mediated amplification)(重要度:★☆☆☆☆)
- NASBA法(nucleic acid sequence-based amplification)(重要度:★☆☆☆☆)
- サザンブロット法・ノーザンブロット法(重要度:★★★★☆)
- ジデオキシ法(DNAシークエンス解析)(重要度:★★★☆☆)
- ISH(in situ hybridization)(重要度:★★★☆☆)
- FISH(fluorescence in situ hybridization)(重要度:★★★☆☆)
- DNAマイクロアレイ法(重要度:★★☆☆☆)
- PCR-RFLP法(polymerase chain reaction-restriction fragment length polymorphism)(重要度:★★☆☆☆)
- 練習問題
遺伝子解析法(重要度:★★★★★)


最近の国家試験のトレンドなのか,遺伝子解析法の問題が増えてきている気がします。
今後もここの範囲は出題が増えてくるはずなので,しっかり押さえておきたいところです。まずは,覚えておきたい遺伝子解析法を示します。
PCR法
リアルタイムPCR法
RT-PCR法・リアルタイムRT-PCR法
LAMP法・TMA法・NASBA法
サザンブロット法・ノーザンブロット法
ジデオキシ法(DNAシークエンス解析)
ISH法・FISH法
DNAマイクロアレイ法
PCR-RFLP法



なお,このページでは簡単な原理しか説明しません。また,使う試薬等も主なものに限っています。
詳細等,興味のある人は各自で調べてみてください。
PCR法(polymerase chain reaction)(重要度:★★★★★)


目的・特徴
特定のDNAの塩基配列を選択的に増幅する方法です。
Nサイクルで約2N倍にDNAを増幅できます。
(20サイクルで約100万倍,30サイクルで約10億倍)
必要なもの
- 鋳型DNA(テンプレートDNA)
増幅させたい配列を持つDNAです。 - プライマー
DNAの増幅開始位置を決定し,相補的なDNAを作る基礎となります。
鋳型DNAのセンス鎖に結合するものをR(reverse)プライマー,鋳型DNAのアンチセンス鎖に結合するものをF(forward)プライマーといいます。 - dNTP(デオキシリボヌクレオシド三リン酸)
d[A/G/C/T]TPのことです。
DNAを作る材料です。それぞれ,DNAの塩基配列のA・G・C・Tとなります。 - DNAポリメラーゼ
dNTPを消費して,DNAを作る酵素です。
現在では高温でも安定で,熱処理でも変性しない耐熱性DNAポリメラーゼが用いられています。 - buffer
- マグネシウムイオン
- サーマルサイクラー
PCRに必要な機械です。
原理
PCRの原理を簡単に説明します。

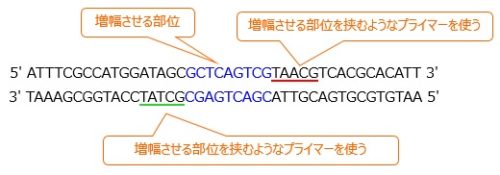
 これが鋳型DNAとします。
これが鋳型DNAとします。
なお,上側がセンス鎖,下側がアンチセンス鎖です。青色の配列が,今回ターゲットとする塩基配列とします。
このとき,プライマーは,このターゲットDNAを挟むような塩基配列をもつものを使用します。
(なお,説明のためプライマーは簡単にしていますが,実際のプライマーの配列はもっと長いです)
なお,図中の赤色・緑色下線は,プライマーが結合する部位を表しています。

1.熱変性(94℃程度)
DNAは2本鎖(dsDNA)なので,まずはこれを1本鎖(ssDNA)にしてDNA増幅を始める準備をします。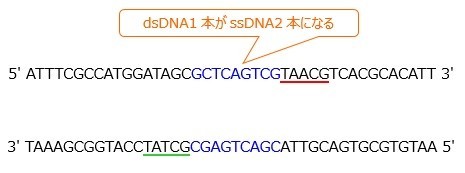
2.アニーリング(60℃程度,プライマーのTm値によって変える)
温度を下げることで,プライマーと鋳型ssDNAが結合して2本鎖を形成します。
※Tm値とは,プライマーがターゲット配列と50%の割合で2本鎖を形成する温度のことです。
このとき,F-プライマーが緑色下線部,R-プライマーが赤色下線部に結合します。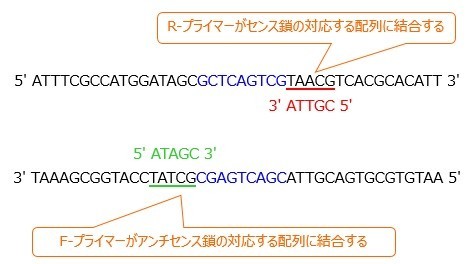
3.伸長反応(72℃程度)
温度を上げてDNAポリメラーゼを活性化させることで,DNAポリメラーゼがDNAを合成します。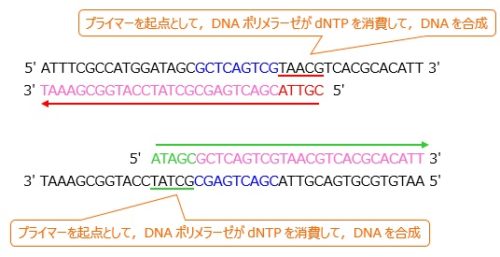
ここまでを1サイクルとして,指定したサイクル数だけ1~3を繰り返します。
PCRで非特異産物ができる/増幅されない
PCRにおいて,増幅量が少ないorまったくバンドが出ない場合の原因と対処法(主なもの)
鋳型DNA濃度が低い。
→基礎となる鋳型DNA濃度を上げる。DNAポリメラーゼが少ない。
→適切な量のDNAポリメラーゼを使う。Mg2+が少ない。
→Mg2+濃度を上げる。PCRサイクル数が少ない。
→サイクル数を多くする。
PCRにおいて,非特異的な増幅産物がみられる場合の原因と対処法(主なもの)
鋳型DNA濃度が高い。
→基礎となる鋳型DNA濃度を下げる。プライマーが過剰である。
→プライマー濃度を低くする。DNAポリメラーゼが多い。
→適切な量のDNAポリメラーゼを使う。Mg2+が多い。
→Mg2+濃度を下げる。アニーリング温度が低すぎるor時間が長すぎる。
→アニーリング温度を上げるor時間を短縮する。PCRサイクル数が多い。
→サイクル数を減らす。
リアルタイムPCR法(real-time polymerase chain reaction)(重要度:★★★☆☆)


目的・特徴
特定のDNAの塩基配列を増幅させ,その増幅を経時的に見る方法です。
Nサイクル後の蛍光強度を測定することで増幅率を計算し,増幅前の鋳型DNAがどれくらいあるかを定量できます。
方法にはインターカレーション法とプローブ法がありますが,ここではプローブ法について説明します。
必要なもの
- 鋳型DNA(テンプレートDNA)
- プライマー
- dNTP(デオキシリボヌクレオシド三リン酸)
- DNAポリメラーゼ
- buffer
- マグネシウムイオン
- サーマルサイクラー

- プローブ
増幅させる塩基配列に特異的な塩基配列を持つものです。
アニーリングの際にプライマーと同様にDNAに結合し,伸長反応によって対象DNAが合成されることで外れます。
プローブには蛍光色素と,蛍光色素の蛍光を抑制する物質(クエンチャー)がそれぞれ結合していて,通常はクエンチャーの作用により蛍光は発しませんが,対象DNAが合成されると蛍光色素とクエンチャーが離れ,蛍光を発するようになります。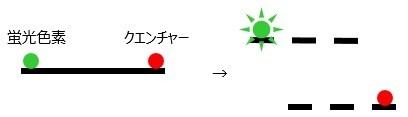
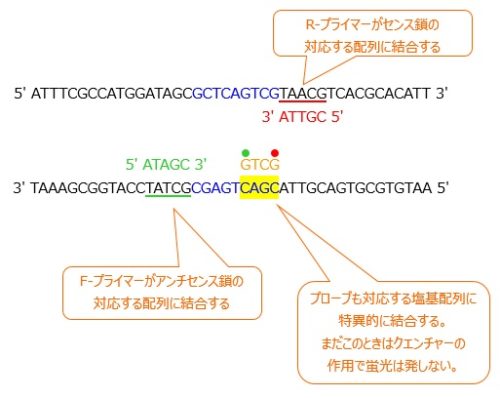
原理
基本的にはPCRの原理と同じです。
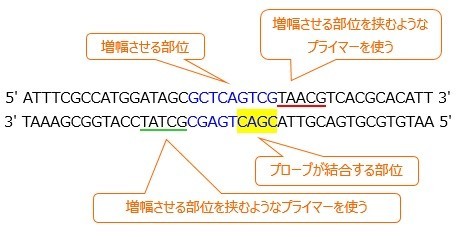
違うところは,アニーリングの際に,プローブも鋳型DNAの特定の配列に結合するというところです。 これが鋳型DNAとします。
これが鋳型DNAとします。
なお,上側がセンス鎖,下側がアンチセンス鎖です。青色の配列が,今回ターゲットとする塩基配列とします。
このとき,プライマーは,このターゲットDNAを挟むような塩基配列をもつものを使用します。
また,プローブも,増幅させる塩基配列の特定の部位を認識するものを使用します。
なお,図中の赤色・緑色下線は,プライマーが結合する部位を表しています。
また,黄色は,プローブが結合する部位を表しています。
1.熱変性(94℃程度)
DNAは2本鎖(dsDNA)なので,まずはこれを1本鎖(ssDNA)にしてDNA増幅を始める準備をします。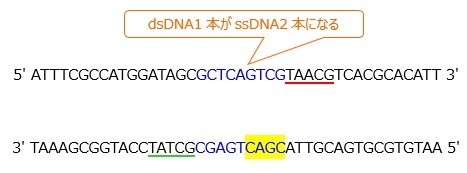
2.アニーリング(60℃程度,プライマーのTm値によって変える)
温度を下げることで,プライマーと鋳型ssDNAが結合して2本鎖を形成します。
このとき,プライマーだけでなく,プローブも特異的な配列に結合します。これをハイブリダイゼーションといいます。
3.伸長反応(72℃程度)
温度を上げてDNAポリメラーゼを活性化させることで,DNAポリメラーゼがDNAを合成します。
このとき,結合したプローブが,DNAポリメラーゼの持つエキソヌクレアーゼ活性によって分解されます。すると,蛍光色素とクエンチャーが離れ,蛍光色素が蛍光を発するようになります。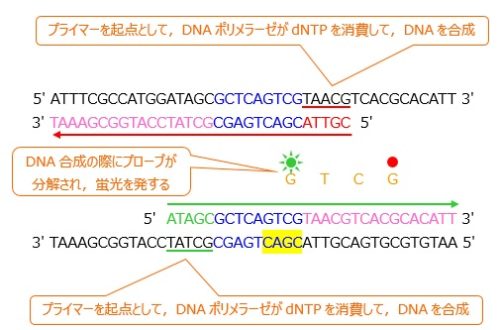
ここまでを1サイクルとして,指定したサイクル数だけ1~3を繰り返します。
サイクルが進むごとに,添加したプローブの分解が進み,蛍光強度が増加します。
RT-PCR法(reverse transcription polymerase chain reaction)(重要度:★★★★☆)



目的・特徴
特定のRNA(細菌のRNAや細胞内に存在するmRNA)の塩基配列を選択的に増幅する方法です。
DNAと違いRNAは1本鎖であり,かつ非常に不安定であるため,通常のPCRでは増やすことができません。そこで,逆転写酵素と呼ばれる,RNAをDNAに変換する酵素を用いてDNAにしてからPCRを行う方法がRT-PCRです。
また,PCR法にリアルタイムがあるように,RT-PCRにもリアルタイム法があります。リアルタイムRT-PCR(RT-qPCR)では,リアルタイムPCRと同じように,増幅前の鋳型RNAがどれくらいあるかを定量できます。
必要なもの
- 鋳型RNA(テンプレートRNA)
- プライマー
- dNTP(デオキシリボヌクレオシド三リン酸)
- DNAポリメラーゼ
- buffer
- マグネシウムイオン
- サーマルサイクラー

逆転写酵素
RNAを鋳型として,RNAと相補的なDNA(complementary DNA,略してcDNA)を合成します。これを逆転写といいます。逆転写用プライマー
逆転写の開始位置を決定し,cDNAを作る基礎となります。
オリゴ(dT)プライマー,ランダムプライマー,遺伝子特異的プライマーの3種類があります。- プローブ
RT-qPCRの場合に用います。
原理
最初に,逆転写酵素と逆転写用プライマーを使って,RNAからcDNAを作製します。
cDNAを作製した後は,PCRの原理に合流します。
LAMP法(loop mediated isothermal amplification)(重要度:★★☆☆☆)
TMA法(transcription mediated amplification)(重要度:★☆☆☆☆)
NASBA法(nucleic acid sequence-based amplification)(重要度:★☆☆☆☆)


目的・特徴
LAMP法
PCRと同様に,特定のDNAの塩基配列を選択的に増幅する方法です。
PCRと違って,温度変化が必要ないことが特徴です。
TMA法
温度変化なしにRNAを増幅する方法です。
RT-PCRが逆転写でcDNAを増幅させるのに比べ,TMAは相補的な一本鎖RNAを増幅させることができる。
NASBA法
TMAと同様,温度変化なしにRNAを増幅する方法です。
RNAを特異的に増幅させることができます。
サザンブロット法・ノーザンブロット法(重要度:★★★★☆)


目的・特徴
特定の塩基配列が存在するかどうかを解析する方法です。
遺伝子を電気泳動で分離し,ゲルから膜(メンブラン)に吸着させ,その後塩基配列に特異的なプローブを加えて,遺伝子とプローブをハイブリダイズさせます。その後,オートラジオグラフィーなどで観察します。
対象がDNAの場合サザンブロット,RNAの場合ノーザンブロットといいます。
必要なもの
DNA/RNA
制限酵素
DNA/RNAを切断するために用います。アガロースゲル
電気泳動で用いるゲルです。ニトロセルロース膜orナイロン膜
電気泳動したDNA/RNAを写し取る(=ブロッティング)のに必要。プローブ
特異的な塩基配列を見つけるために用います。
原理
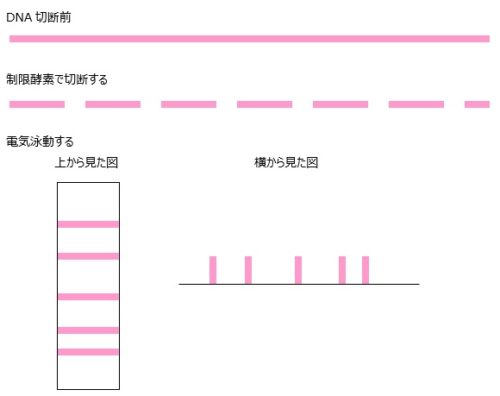
1.制限酵素処理・電気泳動
まず初めにDNA(RNA)を制限酵素で切断し,これを電気泳動します。
2.ブロッティング
次に,これをメンブランと呼ばれる膜に写し取ります(=ブロッティング)。
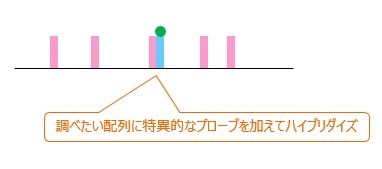
3.プローブをハイブリダイズ
写し取った後,見つけたい塩基配列に特異的なプローブを加えてハイブリダイズさせます。
ハイブリダイズとは,簡単に言うと,プローブとDNA(RNA)をくっつけることです。
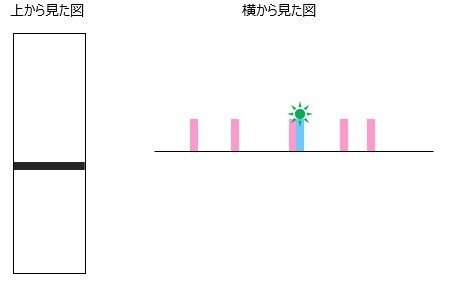
4.オートラジオグラフィなどで可視化
最後に,これをオートラジオグラフィなどで観察します。
ジデオキシ法(DNAシークエンス解析)(重要度:★★★☆☆)
目的・特徴
DNAの塩基配列を調べるために用いる方法です。
必要なもの
基本的にはPCR法と同じです。
最大の違いは,dNTPだけでなく,ddNTPも用いることです。
- 塩基配列を調べたいDNA
- プライマー
- dNTP(デオキシリボヌクレオシド三リン酸)
- DNAポリメラーゼ
- buffer
- マグネシウムイオン
- サーマルサイクラー

- ddNTP(ジデオキシリボヌクレオシド三リン酸)
dd[A/G/C/T]TPのこと。
dNTPの3’末端がOHからHになったものです。ddNTPはdNTPと異なり,これが結合すると,それ以降の伸長反応が起こらなくなります。
=DNA合成反応停止 - 放射性同位体/蛍光色素
放射性同位体を用いる場合,プライマー・dNTP・ddNTPのいずれかに標識を,蛍光色素を用いる場合,ddNTPすべてに標識させます。現在ではその簡便さから,蛍光色素を標識したddNTPを用いる方法が主流です。
原理
以前の方法(放射性同位体標識)
放射性同位体を標識していた場合の方法です。
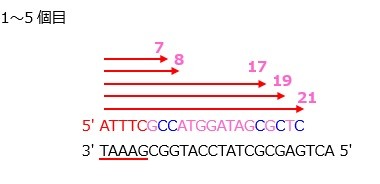
このDNAの塩基配列を調べたいとします。なお,便宜上,3’と5’の配列を逆にしています。
1.前準備
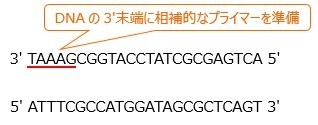
配列を決めたいDNAの3’末端に相補的な短いオリゴヌクレオチドをプライマーとして準備します。
今回の場合,TAAAGの相補鎖であるATTTCがプライマーとなります。
また,ddNTPは,いずれか1種類(例えばddATP)だけを用います。
2.熱変性
DNAは2本鎖(dsDNA)なので,まずはこれを1本鎖(ssDNA)にして塩基配列を調べる準備をします。
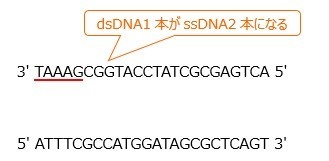
3.アニーリング
温度を下げることで,プライマーと鋳型ssDNAが結合して2本鎖を形成します。
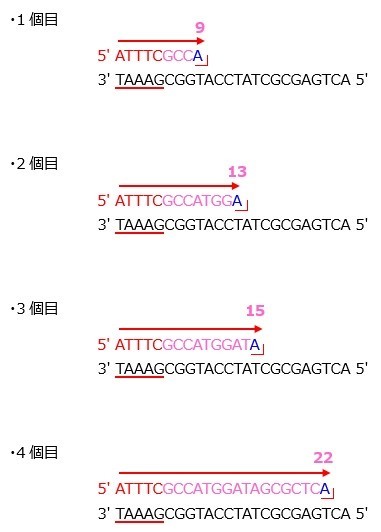
4.伸長反応
温度を上げてDNAポリメラーゼを活性化させることで,DNAポリメラーゼがDNAを合成します。
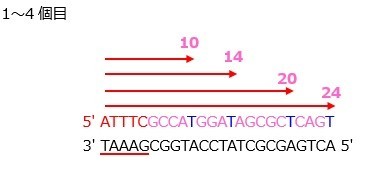
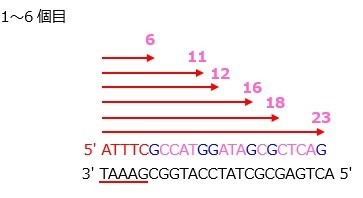
このとき,仮にddATPだけを加えていた場合,Aを合成した段階で反応がストップします。(↓の塩基配列では青色がddNTPを表します)
ただし,ddATPよりも,dATPのほうが多量に存在するため,必ず最初のAではなく,ランダムの位置のAで反応がストップするようになります。
同様にして,ddCTP・ddTTP・ddGTPをそれぞれ1種類ずつ加えた分を用意します。
<ddCTPの場合>
<ddTTPの場合>
<ddGTPの場合>
5.電気泳動・オートラジオグラフィで可視化
これを電気泳動し,オートラジオグラフィで可視化します。
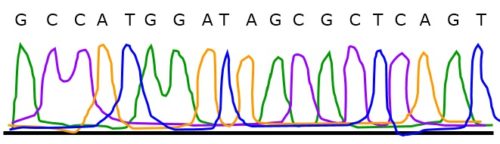
これを,下(分子量の小さいほう)から読んでいくと,以下の配列となります。
読み方としては,バンドがddATPに出ていれば「A」,ddCTPに出ていれば「C」,ddTTPに出ていれば「T」,ddGTPに出ていれば「G」となります。
G C C A T G G A T A G C G C T C A G T
これを,相補鎖に読み替えると目的の配列となります。
C G G T A C C T A T C G C G A G T C A
最後に,プライマー部分の配列の相補鎖を付け加えてシークエンス解析は終了です。
T A A A G C G G T A C C T A T C G C G A G T C A
現在の方法(蛍光標識)
以前の方法は,それぞれのddNTPを1種類だけ加えたものを4種類分用意して,それを4つのレーンに流す方法でしたが,やはり時間がかかるうえに,実験ごとにいちいち放射標識する必要があり面倒でした。
それを改善するため,各ddNTPに違う種類の蛍光色素を標識させることで,1つのサンプルに4つすべてのddNTPを入れることができ,電気泳動で流す数も1レーンで済むようになりました。
原理の1~3は以前の方法と同じです。4の伸長反応は,すべてのddNTPが含まれているため,6~24の大きさの塩基配列が出来上がります。
これを電気泳動すると,各サイズに応じて分離できます。
これにレーザー光を当てると,それぞれの蛍光色素に対応した波長の光が発生します。
これを蛍光検出器で読み取ってコンピュータで処理し,ヒストグラムにします。
このヒストグラムを読むことで,DNA配列を知ることができます。
ISH(in situ hybridization)(重要度:★★★☆☆)
FISH(fluorescence in situ hybridization)(重要度:★★★☆☆)
目的・特徴
組織切片や細胞において,特定のDNAやmRNAの分布や量を検出する方法です。特に,mRNAの検出に用いられます。
ISHはサザンブロットやノーザンブロットと異なり,DNA・RNAの抽出なしで直接組織・細胞にプローブをハイブリダイゼーションさせることができます。
なお,ISHのうち,プローブに蛍光色素を標識させ,蛍光顕微鏡で観察する方法をFISHといいます。
病理の分野でよく用いられる遺伝子検出法です。
免染(免疫染色)と勘違いしやすいですが,免染は蛋白の検出を目的とします。
なお,in situとは,「その場所で」の意味です。
必要なもの
これ以外にもありますが,取り敢えず代表的なもののみを掲載しています。
- 試料(遺伝子発現をみたい組織や細胞)
- プローブ
特異的な塩基配列を見つけるために用います。プローブにはその標識物質の違いにより,放射性標識プローブ,酵素標識プローブ,蛍光標識プローブなどがあります。 - 固定液
4%パラホルムアルデヒド(PFA)などが用いられます。固定をすることで,不安定なmRNAの分解を防止することができます。
原理
サザンブロットやノーザンブロット法と同じで,プローブをハイブリダイズさせて確認します。
DNAマイクロアレイ法(重要度:★★☆☆☆)
目的・特徴
細胞から抽出したmRNAを逆転写酵素でcDNAに変換したものを基板上のDNA配列に対してハイブリダイゼーションすることによって,細胞内で発現している遺伝子情報を網羅的に検出する方法です。
PCR-RFLP法(polymerase chain reaction-restriction fragment length polymorphism)(重要度:★★☆☆☆)


目的・特徴
RFLP(restriction fragment length polymorphism:制限酵素断片長多型)とは,制限酵素によって切断されたDNA断片の長さが,個体間で多型を示すことです。
PCRと組み合わせることで,DNAを増やした後,それを制限酵素で切断して電気泳動することができるようになります。そのときのバンドの出現位置を見ることで,RFLPを見ることができます。
必要なもの
- 鋳型DNA(テンプレートDNA)
- プライマー
- dNTP(デオキシリボヌクレオシド三リン酸)
- DNAポリメラーゼ
- buffer
- マグネシウムイオン
- サーマルサイクラー

- 制限酵素
ある特定の塩基配列で,2本鎖のDNAを切断します。
原理
DNAを増幅するところはPCRと同じです。異なるのは,PCR産物を制限酵素で切断するところからです。
1~3.熱変性→アニーリング→伸長反応
PCR法と同じ
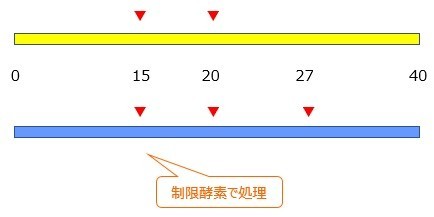
4.制限酵素処理
制限酵素で処理します。今回の場合,▼が制限酵素の働く部分とします。
↓
この制限酵素が働く部分が個体間によって異なるため,切断された断片の長さも異なることになります。これを電気泳動で確認します。
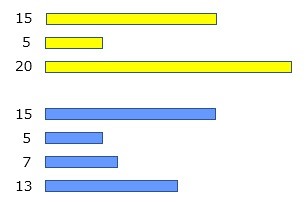
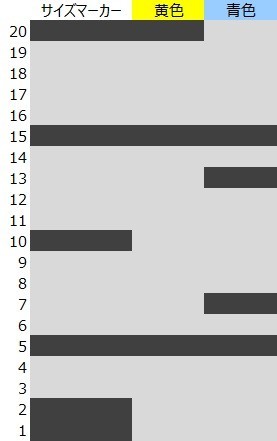
5.電気泳動
電気泳動すると,以下のようなバンドが出現します。なお,サイズマーカーは適当です。
この電気泳動の結果から,制限酵素断片長多型(RFLP)が存在することがわかります。
練習問題

問1 PCR法の反応過程に含まれるのはどれか。2つ選べ。(第61回臨床検査技師国家試験pm10)
1.アルキル化
2.アニーリング
3.トランスダクション
4.ハイブリダイゼーション
5.DNAポリメラーゼ伸長反応
- 解答を見る
解答:2・5
PCR法は「熱変性→アニーリング→伸長反応」の繰り返しです。
問2 PCR法で使用しないのはどれか。(第63回臨床検査技師国家試験pm9)
1.制限酵素
2.プライマー
3.マグネシウムイオン
4.耐熱性DNAポリメラーゼ
5.デオキシリボヌクレオシド三リン酸
- 解答を見る
解答:1
PCR法に制限酵素は使用しません。
制限酵素を使用するのはサザンブロット法やPCR-RFLP法などです。
問3 PCR産物のアガロースゲル電気泳動で非特異的なバンドが見られた場合,対応法として試みるべきなのはどれか。(第65回臨床検査技師国家試験pm9)
1.サイクル数を増やす。
2.Mg2+の濃度を上げる。
3.サンプルの量を増やす。
4.プライマーの量を増やす。
5.アニーリング温度を上げる。
- 解答を見る
解答:5
1~4.これらはPCR産物が全くできなかったときに試みる対処法です。
5.正しい。
問4 プライマーとプローブの両方を用いるのはどれか。(第65回臨床検査技師国家試験am9)
1.FISH法
2.LAMP法
3.ノザンブロット法
4.Real-time PCR法
5.シークエンス解析(ジデオキシ法)
- 解答を見る
解答:4
1.プライマーは不要です。
2.プローブは不要です。
3.プライマーは不要です。
4.正しい。
5.プローブは不要です。
問5 1本鎖cDNA合成に必要なのはどれか。2つ選べ。(第60回臨床検査技師国家試験am44)
1.制限酵素
2.鋳型DNA
3.逆転写酵素
4.DNAポリメラーゼ
5.オリゴ(dT)プライマー
- 解答を見る
解答:3・5
cDNA合成に必要なのは,鋳型となるRNA,逆転写酵素,そしてプライマー(オリゴ(dT)プライマーやランダムプライマーなど)の3つです。よって,3・5が正解となります。
問6 DNAのシークエンス解析に用いないのはどれか。(第62回臨床検査技師国家試験am9)
1.プライマー
2.逆転写酵素
3.DNAポリメラーゼ
4.デオキシヌクレオシド三リン酸
5.蛍光標識ジデオキシヌクレオシド三リン酸
- 解答を見る
解答:2
DNAシークエンス解析(ジデオキシ法)では,PCRと同様に,プライマー・DNAポリメラーゼ・dNTP,そしてシークエンス解析限定で必要な蛍光標識ddNTPが必要となります。よって,2が正解となります。(RNAを扱わないので逆転写酵素は不要です)
問7 正しい組み合わせはどれか。(第53回臨床検査技師国家試験am77)
1.PCR法=DNAの増幅
2.FISH法=蛋白質の解析
3.サザンブロット法=mRNAの解析
4.ノーザンブロット法=DNAの解析
5.ウエスタンブロット法=遺伝子の局在解析
- 解答を見る
解答:1
1.正しい。
2.FISH法は遺伝子発現の解析(特異的塩基配列の有無)を目的とします。
3・4.目的が逆です。
5.ウエスタンブロットは蛋白の解析を目的とします。
問8 遺伝子検査とその目的の組合せで正しいのはどれか。2つ選べ。(第58回臨床検査技師国家試験am44)
1.RT-PCR法=DNAの定量
2.RFLP法=遺伝子多型の解析
3.サザンブロット法=mRNAの解析
4.ノーザンブロット法=DNAの解析
5.in situ ハイブリダイゼーション法=組織での遺伝子発現の局在
- 解答を見る
解答:2・5
1.RT-PCR法はRNAの定量に用いられます。
2.正しい。
3・4.目的が逆です。
5.正しい。
問9 遺伝子検査法とその目的の組合せで誤っているのはどれか。(第63回臨床検査技師国家試験am10)
1.ノーザンブロット法=DNA断片の解析
2.FISH法=欠失解析
3.DNAマイクロアレイ法=網羅的遺伝子解析
4.シークエンス解析法=変異解析
5.マイクロサテライトDNA解析法=キメリズム解析
- 解答を見る
解答:1
1.ノーザンブロット法はRNAの解析が目的です。
2~5.正しい。



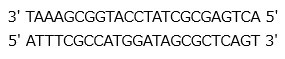





コメント